Jo, Winston Ewerts avhengighetsgraf er en ekte modell
Emily Reeves, 9. august 2022. Oversatt herfra.
Bilde 1. Gibbon-ape
 I mitt forrige innlegg som svar på YouTuber Gutsick Gibbon, alias Erika, lenket jeg til Winston Ewerts 2018 BIO-Complexity-artikkel, "The Dependency Graph of Life", som en mer teknisk forklaring på hvordan vanlig design kan gi et datasett med en tre-lignende struktur. Winstons sentrale tese er at det nestede hierarkiske mønsteret observert i undergrupper av gener er bedre redegjort for, av en avhengighetsgraf som gjenspeiler det faktum at programmerere gjenbruker lignende kodemoduler i forskjellige uavhengige systemer for å oppfylle lignende funksjonelle behov. Erika undersøkte dette litt, og gravde opp en Peaceful Science-dialog og Todd Woods blogg -lenke. Ved 16:02 sier hun:
I mitt forrige innlegg som svar på YouTuber Gutsick Gibbon, alias Erika, lenket jeg til Winston Ewerts 2018 BIO-Complexity-artikkel, "The Dependency Graph of Life", som en mer teknisk forklaring på hvordan vanlig design kan gi et datasett med en tre-lignende struktur. Winstons sentrale tese er at det nestede hierarkiske mønsteret observert i undergrupper av gener er bedre redegjort for, av en avhengighetsgraf som gjenspeiler det faktum at programmerere gjenbruker lignende kodemoduler i forskjellige uavhengige systemer for å oppfylle lignende funksjonelle behov. Erika undersøkte dette litt, og gravde opp en Peaceful Science-dialog og Todd Woods blogg -lenke. Ved 16:02 sier hun:
-I utgangspunktet foreslår [Winston] at livets tre er en slags illusjon av livets tre, og at det faktisk er mer beslektet med å oppdatere programvare, noe som faktisk er ganske merkelig.
Hun fortsetter deretter med å si (19:08):
-Men så er [Winstons modell] egentlig ikke en modell som faktisk kan skilles fra evolusjon, akkurat som intelligent design ikke kan skilles fra evolusjon. Du har litt kryss-symbolikk på gang her.
Bilde 2. Eks. på avhengighetsgraf -Pattedyr
Et distinkt mønster
Men Winstons modell kan skilles fra evolusjon! Som han skrev i sin artikkel som først beskrev avhengighetsgrafmodellen:
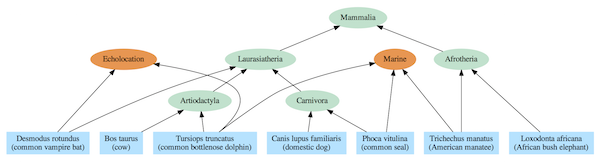 -Avhengighetsgrafen og fellesw anermodeller er forskjellige på ett avgjørende punkt: En art har konvensjonelt én forfedreart, men en modul har vanligvis flere avhengigheter. Se for eksempel Ekkolokalisering og marin i Bilde 2. Alle marine arter er avhengige av en marin modul, og de ekkolokaliserende artene er avhengige av ekkolokaliseringsmodulen. Avhengighetsgrafen er i hovedsak et tre med ekstra fleksibilitet; modulene kan forklare gener som deles mellom arter som antas å være bare fjernt beslektet ved felles avstamning. En modul er ikke begrenset til å gjenbruke kode fra en enkelt kilde, men kan fritt gjenbruke fra flere kilder. Sammenlign dette med felles avstamning der hver art nesten utelukkende må hente fra en enkelt kilde: dens forfedres art.
-Avhengighetsgrafen og fellesw anermodeller er forskjellige på ett avgjørende punkt: En art har konvensjonelt én forfedreart, men en modul har vanligvis flere avhengigheter. Se for eksempel Ekkolokalisering og marin i Bilde 2. Alle marine arter er avhengige av en marin modul, og de ekkolokaliserende artene er avhengige av ekkolokaliseringsmodulen. Avhengighetsgrafen er i hovedsak et tre med ekstra fleksibilitet; modulene kan forklare gener som deles mellom arter som antas å være bare fjernt beslektet ved felles avstamning. En modul er ikke begrenset til å gjenbruke kode fra en enkelt kilde, men kan fritt gjenbruke fra flere kilder. Sammenlign dette med felles avstamning der hver art nesten utelukkende må hente fra en enkelt kilde: dens forfedres art.
Denne modellen forutsier et distinkt mønster fra felles avstamning. Som et eksempel forutsier avhengighetsgrafen at moduler som ikke samsvarer med taksonomiske kategorier (dvs. gjenbruk av komponenter på en måte som ikke passer til et tre) bør være rikelig. I tilfelle du vil lese konklusjonen fra Winstons arbeid:
-Forutsigelsene fra avhengighetsgrafhypotesen satt opp i denne artikkelen har vist seg å være korrekte. De biologiske dataene passet bedre til en avhengighetsgraf enn til et tre. Dataene produsert av en simulert prosess med felles avstamning passet bedre til et tre enn til en avhengighetsgraf. Dataene produsert av en kompilator passet både bedre til en avhengighetsgraf enn et tre, og passet bedre til et tre enn til nullmodellen. De utledede biologiske avhengighetsgrafene inneholdt ikke bare livets tre med noen få tillegg, men inneholdt i stedet mange tilleggsmoduler.
Bilde 3. Gjenbruk av data
Antyder trelignende data i det hele felles aner?
 En interessant observasjon av Erika, opprinnelig reist av beregningsbiolog Joshua Swamidass ved Peaceful Science, er at menneskelig mangfold ikke alltid viser et tremønster. Argumentet er at selv i et tilfelle der vi alle er enige om at felles aner er sann (dvs. innen mennesker), finner vi ikke nødvendigvis et trelignende datasett. Dette er ment å få felles avstamning 'bort fra kroken', så det ikke blir forfalsket eller utfordret i de mange andre tilfellene, der dataene ikke er trelignende. Et par punkter som svar:
En interessant observasjon av Erika, opprinnelig reist av beregningsbiolog Joshua Swamidass ved Peaceful Science, er at menneskelig mangfold ikke alltid viser et tremønster. Argumentet er at selv i et tilfelle der vi alle er enige om at felles aner er sann (dvs. innen mennesker), finner vi ikke nødvendigvis et trelignende datasett. Dette er ment å få felles avstamning 'bort fra kroken', så det ikke blir forfalsket eller utfordret i de mange andre tilfellene, der dataene ikke er trelignende. Et par punkter som svar:
1. Felles avstamning har historisk sett forutsagt et trelignende mønster. Hvis den ikke gjør det, hvorfor evaluerer praktisk talt alle fylogenetikkstudier konsistensindeksen - dvs. hvorfor rapporterer den statistikk som forteller oss hvor 'trelignende' et datasett er? Videre, hvis felles aner ikke forutsier en slags tre, hvorfor foretrekker da fylogenetiske studier alltid å rapportere det mest 'trelignende' mønsteret for å beskrive datasettet deres som det minst omfattende 'treet', antydet av dataene? Hele premisset til Baum et al. (2016) er at felles avstamning er en bedre forklaring, fordi den ser ut til å være mer treaktig enn tilfeldig i utbredelsen.
2. Nå er det sant at det er kjente prosesser i biologi som kan produsere ikke-trelignende mønstre, men det er fortsatt slik at jo mer trelignende et datasett er, jo mer tillit har vi til at det ble produsert av felles aner. Mange datasett kan lande i gråsonen der skeptikere kan føle at de er for lite trelignende til å bli produsert ved felles avstamning, og 'troende' om felles avstamning, føler at de er for treaktige til å bli produsert med felles design. Mange samtaler gjenstår, men uansett hva svaret er, trenger vi realistiske vurderinger av hva designbaserte modeller vil forutsi. Jeg håper dette innlegget har bidratt til å bringe samtalen videre på en positiv måte.
Før jeg avslutter denne serien i morgen, vil jeg avslutte med dette for nå: Det faktum at kjente naturlige biologiske prosesser kan produsere ikke-tre-lignende mønstre, er problematisk ikke for ID-tilhengere, men for tilhengere av felles avstamning. Hvis vi, når vi vet at genetisk arv forekommer, ikke ser et tremønster, hva sier dette om vår evne til å teste eller demonstrere felles aner? Tilstedeværelsen av ikke-trelignende data er imidlertid ikke et problem for ID-tilhengere som utfordrer felles aner. Det er fordi vi føler at vi kan forklare genetisk likhet bedre gjennom felles design.
La oss drive forutsetningsfri vitenskap! Noen avsluttende tanker om Gutsick Gibbons utfordring
Emily Reeves; 10. august Oversatt herfra.
Bilde 4. Sml. med ingeniør-baserte systemer
 I en serie med innlegg (her og her) har jeg svart YouTuber Gutsick Gibbon, aka Erika, på spørsmålet om artikkelen til Baum et al. (2016) foreslo en rimelig test av den separate anermodellen. Jeg håper jeg har overbevist deg, min leser, om at de ikke gjorde det. Deres krav om at separate aner involverer tilfeldige fordelinger av egenskaper, har ikke blitt foreslått av ID-tilhengere og er i strid med hvordan menneskelige designere designer ting.
I en serie med innlegg (her og her) har jeg svart YouTuber Gutsick Gibbon, aka Erika, på spørsmålet om artikkelen til Baum et al. (2016) foreslo en rimelig test av den separate anermodellen. Jeg håper jeg har overbevist deg, min leser, om at de ikke gjorde det. Deres krav om at separate aner involverer tilfeldige fordelinger av egenskaper, har ikke blitt foreslått av ID-tilhengere og er i strid med hvordan menneskelige designere designer ting.
De statistiske nullene i Baum et al. (2016), som Erika selv erklærte "Sprøtt!" -lenke, er ikke et bevis for usannsynligheten for faktiske separate aner, men på den dårlige utformingen av deres test av den separate anermodellen.
Blanding av funksjonelle synapomorfier/trekkdata som grupperer seg på grunn av optimalisering eller begrensninger er ikke en gyldig modell for separate aner. Det er fordi det strider mot alt vi vet om hvordan design fungerer. Designet teknologi inneholder komponenter med korrelasjoner og mønstre som, når de sees gjennom en fylogenetisk linse, kan gi en viss grad av trelignende data. Det betyr ikke at de oppsto på grunn av felles aner. Det betyr at de oppsto på grunn av felles design for å møte funksjonelle krav.
Det ID-tilhengere oppmuntrer i det større vitenskapelige miljøet er ærlighet om det faktum at både design og aner kan skape genetisk likhet. Vi sier at alle må tenke annerledes om det hierarkiske signalet vi observerer i livet. I et nøtteskall, la oss begynne å gjøre antagelsesfri vitenskap innen fylogenetikk der man ikke på forhånd utelukker design som en rimelig hypotese.
Som jeg har merket, stiger eller faller ikke intelligent design med felles aner, for ID-tilhengere har forskjellige syn på det spørsmålet. Jeg stoler imidlertid på at jeg har gjort det helt klart at ID-tilhengere og andre som stiller spørsmål ved felles aner har utmerkede vitenskapelige grunner for å gjøre det.
 Emily Reeves
Emily Reeves
Emily Reeves er en biokjemiker, metabolsk ernæringsfysiolog og aspirerende systembiolog. Doktorgradsstudiene hennes ble fullført ved Texas A&M University i biokjemi og biofysikk. Emily er for tiden en aktiv kliniker for metabolsk ernæring og ernæringsgenomikk ved Nutriplexity. Hun liker å identifisere og designe ernæringsintervensjon for subtile medfødte feil i stoffskiftet. Hun jobber også med stipendiater fra Discovery Institute og det større vitenskapelige samfunnet for å fremme integrering av ingeniørvitenskap og biologi. Hun tilbringer helgene sine på eventyr med mannen sin, brygger kombucha og løper i nærheten av Puget Sound.
Bilde 4. Emily Reeves
Ovesettelse og bilder ved Asbjørn E. Lund
Kilder
Baum, David A., Cécile Ané, Bret Larget, Claudia Solís-Lemus, Lam Si Tung Ho, Peggy Boone, Chloe P. Drummond, Martin Bontrager, Steven J.
Hunter, and William Saucier. 2016. “Statistical Evidence for Common Ancestry: Application to Primates.” Evolution: International Journal of Organic Evolution 70 (6): 1354–63.
Doolittle, W. Ford, and Eric Bapteste. 2007. “Pattern Pluralism and the Tree of Life Hypothesis.” Proceedings of the National Academy of Sciences of the United States of America 104 (7): 2043–49.
Ewert, Winston. 2018. “The Dependency Graph of Life.” BIO-Complexity. https://doi.org/10.5048/bio-c.2018.3.
Murphy, W. J., E. Eizirik, W. E. Johnson, Y. P. Zhang, O. A. Ryder, and S. J. O’Brien. 2001. “Molecular Phylogenetics and the Origins of Placental Mammals.” Nature 409 (6820): 614–18.
Perelman, Polina, Warren E. Johnson, Christian Roos, Hector N. Seuánez, Julie E. Horvath, Miguel A. M. Moreira, Bailey Kessing, et al. 2011. “A Molecular Phylogeny of Living Primates.” PLOS Genetics 7 (3): e1001342.
 I mitt forrige innlegg som svar på YouTuber Gutsick Gibbon, alias Erika, lenket jeg til Winston Ewerts 2018 BIO-Complexity-artikkel, "The Dependency Graph of Life", som en mer teknisk forklaring på hvordan vanlig design kan gi et datasett med en tre-lignende struktur. Winstons sentrale tese er at det nestede hierarkiske mønsteret observert i undergrupper av gener er bedre redegjort for, av en avhengighetsgraf som gjenspeiler det faktum at programmerere gjenbruker lignende kodemoduler i forskjellige uavhengige systemer for å oppfylle lignende funksjonelle behov. Erika undersøkte dette litt, og gravde opp en Peaceful Science-dialog og Todd Woods blogg -lenke. Ved 16:02 sier hun:
I mitt forrige innlegg som svar på YouTuber Gutsick Gibbon, alias Erika, lenket jeg til Winston Ewerts 2018 BIO-Complexity-artikkel, "The Dependency Graph of Life", som en mer teknisk forklaring på hvordan vanlig design kan gi et datasett med en tre-lignende struktur. Winstons sentrale tese er at det nestede hierarkiske mønsteret observert i undergrupper av gener er bedre redegjort for, av en avhengighetsgraf som gjenspeiler det faktum at programmerere gjenbruker lignende kodemoduler i forskjellige uavhengige systemer for å oppfylle lignende funksjonelle behov. Erika undersøkte dette litt, og gravde opp en Peaceful Science-dialog og Todd Woods blogg -lenke. Ved 16:02 sier hun: -Avhengighetsgrafen og fellesw anermodeller er forskjellige på ett avgjørende punkt: En art har konvensjonelt én forfedreart, men en modul har vanligvis flere avhengigheter. Se for eksempel Ekkolokalisering og marin i Bilde 2. Alle marine arter er avhengige av en marin modul, og de ekkolokaliserende artene er avhengige av ekkolokaliseringsmodulen. Avhengighetsgrafen er i hovedsak et tre med ekstra fleksibilitet; modulene kan forklare gener som deles mellom arter som antas å være bare fjernt beslektet ved felles avstamning. En modul er ikke begrenset til å gjenbruke kode fra en enkelt kilde, men kan fritt gjenbruke fra flere kilder. Sammenlign dette med felles avstamning der hver art nesten utelukkende må hente fra en enkelt kilde: dens forfedres art.
-Avhengighetsgrafen og fellesw anermodeller er forskjellige på ett avgjørende punkt: En art har konvensjonelt én forfedreart, men en modul har vanligvis flere avhengigheter. Se for eksempel Ekkolokalisering og marin i Bilde 2. Alle marine arter er avhengige av en marin modul, og de ekkolokaliserende artene er avhengige av ekkolokaliseringsmodulen. Avhengighetsgrafen er i hovedsak et tre med ekstra fleksibilitet; modulene kan forklare gener som deles mellom arter som antas å være bare fjernt beslektet ved felles avstamning. En modul er ikke begrenset til å gjenbruke kode fra en enkelt kilde, men kan fritt gjenbruke fra flere kilder. Sammenlign dette med felles avstamning der hver art nesten utelukkende må hente fra en enkelt kilde: dens forfedres art. En interessant observasjon av Erika, opprinnelig reist av beregningsbiolog Joshua Swamidass ved Peaceful Science, er at menneskelig mangfold ikke alltid viser et tremønster. Argumentet er at selv i et tilfelle der vi alle er enige om at felles aner er sann (dvs. innen mennesker), finner vi ikke nødvendigvis et trelignende datasett. Dette er ment å få felles avstamning 'bort fra kroken', så det ikke blir forfalsket eller utfordret i de mange andre tilfellene, der dataene ikke er trelignende. Et par punkter som svar:
En interessant observasjon av Erika, opprinnelig reist av beregningsbiolog Joshua Swamidass ved Peaceful Science, er at menneskelig mangfold ikke alltid viser et tremønster. Argumentet er at selv i et tilfelle der vi alle er enige om at felles aner er sann (dvs. innen mennesker), finner vi ikke nødvendigvis et trelignende datasett. Dette er ment å få felles avstamning 'bort fra kroken', så det ikke blir forfalsket eller utfordret i de mange andre tilfellene, der dataene ikke er trelignende. Et par punkter som svar: I en serie med innlegg (her og her) har jeg svart YouTuber Gutsick Gibbon, aka Erika, på spørsmålet om artikkelen til Baum et al. (2016) foreslo en rimelig test av den separate anermodellen. Jeg håper jeg har overbevist deg, min leser, om at de ikke gjorde det. Deres krav om at separate aner involverer tilfeldige fordelinger av egenskaper, har ikke blitt foreslått av ID-tilhengere og er i strid med hvordan menneskelige designere designer ting.
I en serie med innlegg (her og her) har jeg svart YouTuber Gutsick Gibbon, aka Erika, på spørsmålet om artikkelen til Baum et al. (2016) foreslo en rimelig test av den separate anermodellen. Jeg håper jeg har overbevist deg, min leser, om at de ikke gjorde det. Deres krav om at separate aner involverer tilfeldige fordelinger av egenskaper, har ikke blitt foreslått av ID-tilhengere og er i strid med hvordan menneskelige designere designer ting. Emily Reeves
Emily Reeves